Автор: Денис Аветисян
Исследователи предлагают принципиально новый подход к моделированию взаимодействия лекарственных препаратов, основанный на обобщении знаний о связях между молекулами.
Пока крипто-инвесторы ловят иксы и ликвидации, мы тут скучно изучаем отчетность и ждем дивиденды. Если тебе близка эта скука, добро пожаловать.
Купить акции "голубых фишек"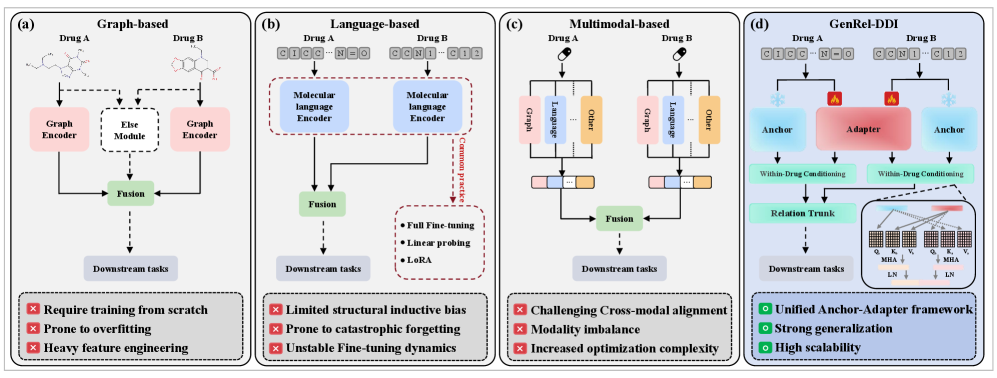
Предложенная модель GenRel-DDI обеспечивает передовую производительность в задачах прогнозирования взаимодействий, особенно в сложных сценариях с разделением сущностей, благодаря отделению молекулярных представлений от паттернов взаимодействия.
Несмотря на значительные успехи в предсказании лекарственных взаимодействий, существующие модели часто демонстрируют низкую обобщающую способность в реальных сценариях, особенно при работе с новыми лекарственными препаратами. В данной работе, озаглавленной ‘Rethinking Drug-Drug Interaction Modeling as Generalizable Relation Learning’, предложен новый подход GenRel-DDI, который переосмысливает задачу предсказания взаимодействий как обучение отношениям, а не свойствам отдельных молекул. Это позволяет отделить представление о взаимодействии от идентификаторов лекарств и добиться существенного улучшения обобщающей способности, особенно в условиях строгих ограничений на пересечение данных. Сможет ли подобный подход стать основой для создания более надежных и эффективных систем прогнозирования лекарственных взаимодействий в клинической практике?
Проблема прогнозирования лекарственных взаимодействий
В связи с растущей распространенностью полипрагмазии — одновременного приема множества лекарственных препаратов — точное прогнозирование лекарственных взаимодействий (ЛВ) приобретает первостепенное значение для безопасности пациентов. Комбинирование нескольких медикаментов значительно повышает вероятность нежелательных реакций, которые могут варьироваться от незначительных побочных эффектов до серьезных осложнений, требующих немедленной медицинской помощи. Поэтому, разработка надежных методов прогнозирования ЛВ является критически важной задачей современной фармакологии и клинической практики, позволяющей минимизировать риски и оптимизировать терапевтические результаты для пациентов, вынужденных принимать несколько препаратов одновременно. Неспособность предвидеть такие взаимодействия может привести к снижению эффективности лечения, увеличению затрат на здравоохранение и, в конечном итоге, к ухудшению качества жизни.
Существующие методы предсказания лекарственных взаимодействий (ЛВ) зачастую демонстрируют ограниченную способность к обобщению, что представляет серьезную угрозу для безопасности пациентов. Традиционные подходы, как правило, хорошо работают с известными комбинациями препаратов, но их эффективность резко снижается при оценке взаимодействий, которые ранее не встречались в обучающих данных. Это связано с тем, что ЛВ — сложные процессы, зависящие от множества факторов, включая фармакокинетику, фармакодинамику и индивидуальные особенности организма. Неспособность адекватно прогнозировать эффекты от новых комбинаций лекарств может привести к нежелательным побочным реакциям, снижению эффективности лечения и даже летальному исходу, подчеркивая необходимость разработки более надежных и адаптивных методов предсказания ЛВ.
Сложность лекарственных взаимодействий требует разработки инновационных подходов, способных к обучению на основе данных, выходящих за рамки известных комбинаций препаратов. Традиционные методы часто ограничены известными взаимодействиями, что не позволяет предсказать эффекты от новых или редких сочетаний лекарств. Для преодоления этой проблемы исследователи обращаются к алгоритмам машинного обучения, способным выявлять скрытые закономерности в больших объемах данных, включая геномные профили пациентов и молекулярные характеристики препаратов. Такие подходы позволяют предсказывать взаимодействия, основываясь не только на известных фактах, но и на вероятностных моделях, учитывающих индивидуальные особенности организма и сложные химические процессы, происходящие в ходе метаболизма лекарств. Это открывает перспективы для персонализированной фармакологии и повышения безопасности полифармакотерапии.
Новый взгляд на прогнозирование взаимодействий: реляционное обучение
GenRel-DDI представляет новую структуру, отделяющую идентификацию лекарственного препарата от предсказания его взаимодействия. В отличие от традиционных методов, которые полагаются на специфические свойства каждого препарата, GenRel-DDI позволяет обобщать данные и прогнозировать взаимодействие с ранее не встречавшимися лекарственными средствами. Это достигается за счет фокусировки на общих принципах, определяющих взаимодействие между препаратами, а не на их индивидуальных характеристиках, что повышает эффективность предсказания при работе с новыми лекарствами и снижает зависимость от полных данных о каждом препарате.
В основе подхода GenRel-DDI лежит концепция реляционного обучения, позволяющая моделировать фундаментальные принципы взаимодействия лекарственных средств, а не полагаться на специфические свойства каждого препарата. Традиционные методы часто фокусируются на характеристиках молекул, что ограничивает обобщающую способность модели при работе с новыми соединениями. Реляционное обучение, напротив, стремится выявить общие закономерности, определяющие взаимодействие между препаратами, независимо от их индивидуальных особенностей. Это достигается путем представления взаимодействий как отношений между лекарствами и моделирования этих отношений с помощью нейронных сетей, что позволяет предсказывать эффекты новых комбинаций препаратов, основываясь на изученных принципах взаимодействия, а не на конкретных свойствах входящих в них веществ.
Архитектура «Якорь-Адаптер» представляет собой ключевой компонент предложенного подхода. Она включает в себя фиксированный, предварительно обученный энкодер, отвечающий за извлечение общих признаков из молекулярных представлений лекарственных средств, и обучаемый реляционный модуль. Этот модуль специализируется на моделировании взаимосвязей между лекарствами, позволяя системе обобщать знания на новые, ранее не встречавшиеся соединения. Фиксированный энкодер обеспечивает стабильное представление базовых характеристик лекарств, в то время как обучаемый модуль адаптируется к конкретным типам взаимодействий, обеспечивая гибкость и точность предсказаний.
Кодирование лекарственных средств и их взаимодействий
В GenRel-DDI для создания устойчивых представлений молекул используются предварительно обученные энкодеры, такие как MolT5, MoLFormer и ChemBERTa-3. Эти модели, обученные на больших объемах химических данных, способны извлекать и кодировать значимые признаки молекулярной структуры и свойств. MolT5, основанный на архитектуре Transformer, эффективно преобразует молекулярные графы в последовательности токенов. MoLFormer использует механизм внимания для выявления важных взаимодействий между атомами в молекуле. ChemBERTa-3, являясь вариантом BERT, адаптирован для химических данных и обеспечивает контекстуализированные представления молекул. Использование предварительно обученных моделей позволяет значительно улучшить качество представлений и, как следствие, повысить точность предсказания взаимодействий между лекарственными средствами.
В GenRel-DDI, полученные предварительно обученными энкодерами представления молекул объединяются (fusion) в рамках общей архитектуры для предсказания взаимодействий между ними. Этот процесс позволяет эффективно улавливать сложные химические взаимосвязи, поскольку объединение представлений позволяет модели учитывать различные аспекты структуры и свойств молекул при определении вероятности и характера их взаимодействия. Используемые методы fusion направлены на выделение наиболее релевантных признаков из отдельных представлений для точного прогнозирования.
Молекулярно-ориентированный конвейер (Molecule-Centric Pipeline) представляет собой базовый подход к кодированию лекарственных средств перед анализом их взаимодействий. Он предполагает предварительную обработку и преобразование информации о структуре молекул в векторные представления, которые затем используются для выявления потенциальных взаимодействий между препаратами. Этот этап позволяет стандартизировать входные данные и выделить ключевые характеристики молекул, необходимые для точного прогнозирования эффектов взаимодействия, что повышает надежность последующего анализа и моделирования.
Надежная оценка и обобщающая способность
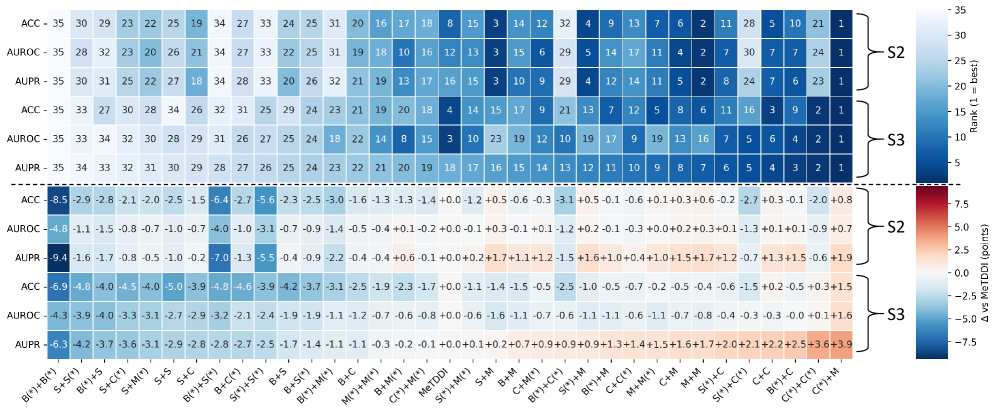
Для обеспечения надежной оценки, разработанный фреймворк тестируется на стандартных наборах данных, таких как MeTDDI и DDInter. При этом используется стратегия разделения данных с контролем перекрытия (overlap-controlled splits), позволяющая избежать завышения результатов за счет попадания схожих объектов в обучающую и тестовую выборки. Данный подход гарантирует, что оценка производительности отражает реальную способность модели обобщать знания на новые, ранее не встречавшиеся данные, а не просто запоминать характеристики обучающей выборки.
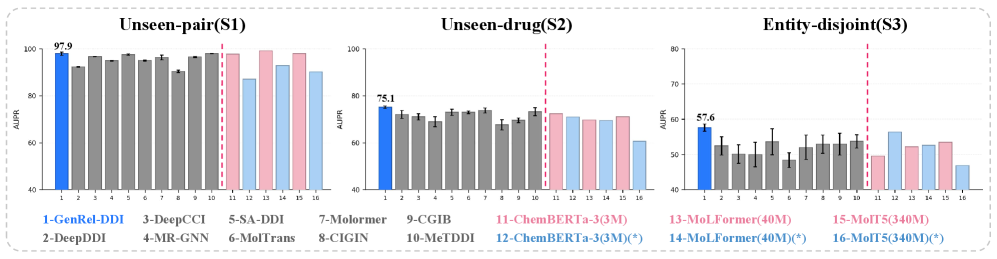
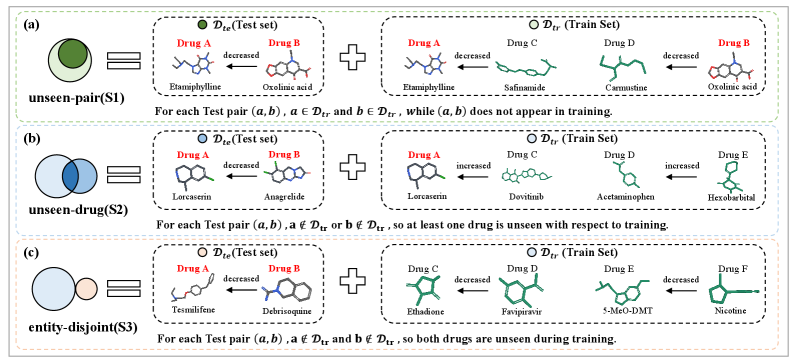
Для всесторонней оценки способности модели к обобщению используются специализированные наборы данных, разделенные на несколько типов. Наборы данных с разделением «Unseen-Pair» содержат пары лекарственных препаратов и мишеней, которые не встречаются в обучающей выборке. В наборах «Unseen-Drug» используются лекарственные препараты, отсутствующие в обучающей выборке, а «Entity-Disjoint» разделяют обучающие и тестовые данные по принципу полного отсутствия общих сущностей (лекарств или мишеней). Такой подход позволяет оценить, насколько хорошо модель предсказывает взаимодействия для новых, ранее не встречавшихся комбинаций лекарств и мишеней, обеспечивая более реалистичную оценку её производительности в реальных условиях.
Набор данных MeTDDI S1 использовался для оценки производительности GenRel-DDI, в результате чего была достигнута выдающаяся Area Under the Receiver Operating Characteristic curve (AUROC) в 99.3%. Данный показатель свидетельствует о высокой точности предсказаний модели в отношении выявления взаимодействий между лекарственными препаратами и заболеваниями в рамках данного набора данных. Высокое значение AUROC указывает на способность модели эффективно различать истинные взаимодействия от ложных, что является критически важным для обеспечения безопасности пациентов и эффективности лечения.
В ходе оценки на наборе данных MeTDDI S3, модель GenRel-DDI продемонстрировала улучшение показателя AUPR на 3.9% по сравнению с базовым уровнем. Данное увеличение свидетельствует о повышенной способности модели предсказывать взаимодействия в сложных сценариях, где стандартные подходы могут оказаться менее эффективными. Повышение AUPR указывает на улучшенное обнаружение истинно положительных результатов при сохранении низкого уровня ложноположительных срабатываний, что критически важно для надежности предсказаний взаимодействий.
В ходе оценки на общедоступных бенчмарках DeepDDI, ZhangDDI и ChChDDI, предложенная система продемонстрировала наивысшие зарегистрированные показатели AUROC (Area Under the Receiver Operating Characteristic curve) и AUPR (Area Under the Precision-Recall curve). Данный результат подтверждает, что система достигла передового уровня производительности в задаче предсказания взаимодействий лекарственных средств и химических соединений, превосходя существующие аналоги по данным метрикам на указанных наборах данных.
Представленная работа демонстрирует элегантный подход к моделированию взаимодействий лекарственных препаратов, рассматривая задачу как обучение отношениям. Это позволяет отделить молекулярные представления от паттернов взаимодействия, что особенно ценно в сложных сценариях, где требуется обобщение на новые сущности. Как отмечал Эдсгер Дейкстра: «Простота — это главное. Начните с простого и добавляйте сложность только тогда, когда это необходимо». Этот принцип находит отражение в архитектуре GenRel-DDI, где стремление к ясности структуры способствует повышению обобщающей способности модели и, следовательно, ее надежности в предсказании взаимодействий лекарств.
Куда двигаться дальше?
Представленная работа, фокусируясь на обобщающей способности моделей предсказания лекарственных взаимодействий, поднимает вопрос о фундаментальной природе этих взаимодействий. Если взаимодействие определяется лишь сопоставлением признаков молекул, то, по сути, мы имеем дело с очень сложной, но всё же таблицей соответствий. Иллюзия контроля над системой возникает, когда модульность не подкрепляется глубоким пониманием контекста — простое добавление слоёв внимания не устраняет необходимость в ясной, принципиальной модели. Если система держится на костылях из сложных ансамблей, значит, мы переусложнили её, пытаясь описать слишком много, не понимая сути.
Особый интерес вызывает устойчивость предложенного подхода к сценариям, где новые молекулы существенно отличаются от тех, на которых обучалась модель. Обобщение — это не просто достижение высокой точности на отложенной выборке, но и способность адаптироваться к принципиально новым условиям. Необходимо исследовать, как предложенный фреймворк справляется с взаимодействиями, обусловленными не столько химической структурой, сколько специфическими биологическими механизмами, которые могут быть не отражены в исходных данных.
Будущие исследования должны сосредоточиться на интеграции знаний из различных источников — геномики, протеомики, метаболомики — для создания более полной и реалистичной модели лекарственных взаимодействий. Успех в этой области потребует не только улучшения алгоритмов машинного обучения, но и развития фундаментального понимания биологических процессов, лежащих в основе этих взаимодействий. Простая точность — недостаточна; требуется элегантность и ясность.
Оригинал статьи: https://arxiv.org/pdf/2601.15771.pdf
Связаться с автором: https://www.linkedin.com/in/avetisyan/
Смотрите также:
- Российский рынок: Оптимизм на фоне трехсторонних переговоров – чего ждать 1 февраля? (25.01.2026 17:32)
- Сургутнефтегаз акции привилегированные прогноз. Цена SNGSP
- Новые смартфоны. Что купить в январе 2026.
- Российская экономика 2025: Рекорды энергопотребления, падение добычи и укрепление рубля (22.01.2026 17:32)
- Российский рынок: Оптимизм на фоне геополитики и корпоративных сделок (20.01.2026 00:32)
- Cubot Note 60 ОБЗОР: плавный интерфейс, большой аккумулятор
- Что такое виньетирование? Коррекция периферийного освещения в Кэнон.
- Lava Agni 4 ОБЗОР: большой аккумулятор, яркий экран, плавный интерфейс
- Типы дисплеев. Какой монитор выбрать?
- Xiaomi Redmi A3 Pro ОБЗОР: большой аккумулятор, удобный сенсор отпечатков
2026-01-25 04:14
